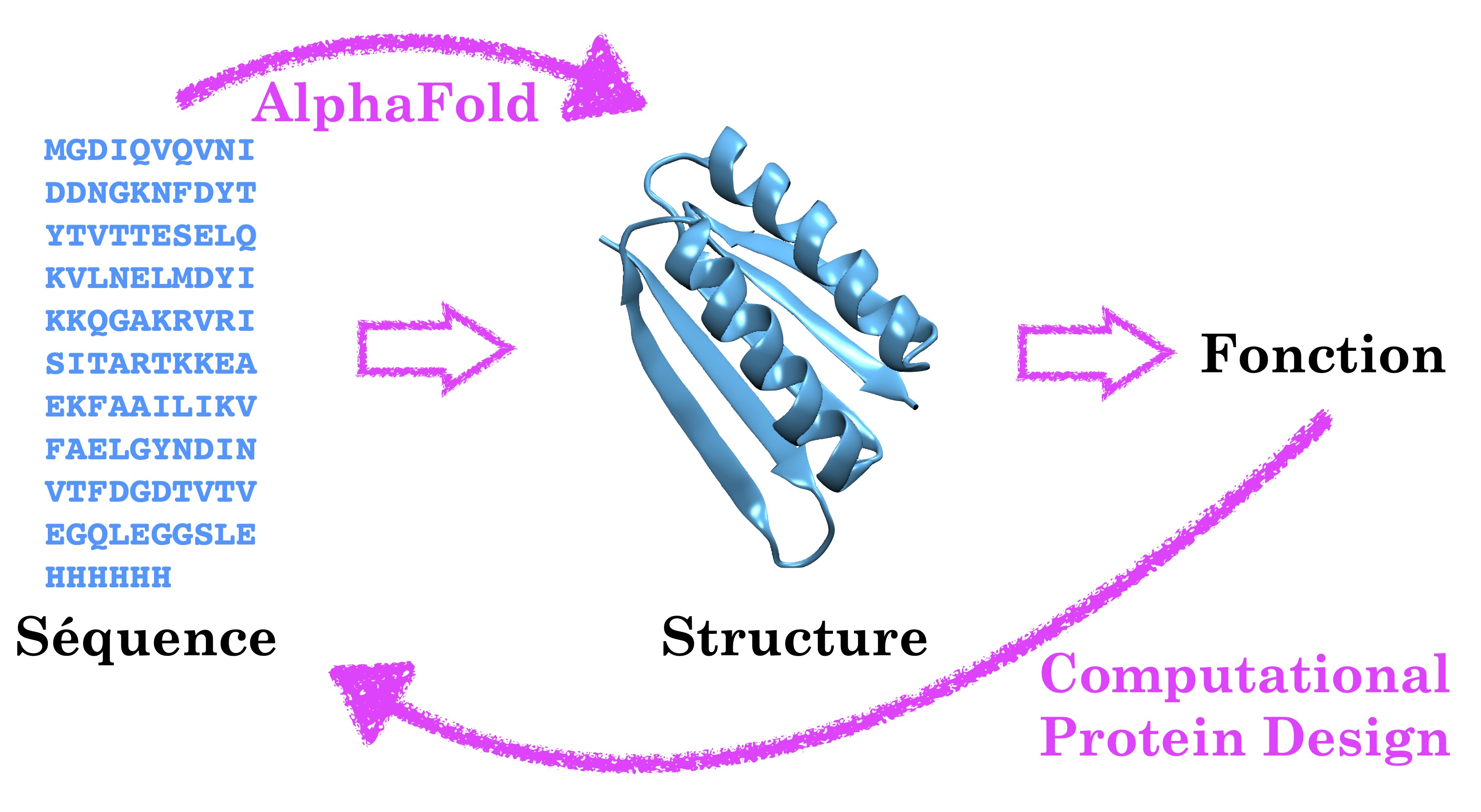
En octobre 2024, le Prix Nobel de Chimie a été attribué pour moitié à l’équipe responsable du développement du logiciel AlphaFold2, pour son éclatante réussite en terme de prédiction de structure des protéines. L’autre moitié du prix est revenue à David Baker, un chercheur certes moins connu du grand public, mais pourtant une star de sa communauté scientifique. Depuis plus de trente ans, les travaux de son équipe de recherche ont permis des avancées considérables dans notre compréhension de la façon dont la séquence d’une protéine va déterminer sa structure, et par conséquence sa fonction au sein de la cellule. Mais cela a également amené l’équipe de David Baker à résoudre le problème inverse. À savoir, si l’on part d’une fonction protéique particulière, quelle serait la structure, et même la séquence d’acides aminés qui permettraient d’obtenir cette fonction ?
Quand ce problème est abordé via des outils de modélisation informatique, on parle de Computational Protein Design (CPD). Et ce sont les réussites de l’équipe Baker dans le domaine qui lui ont valu une moitié de Nobel le mois dernier. Leur premier succès en la matière date de 2003, avec la conception de TOP7, une petite protéine artificielle (dite aussi de novo) d’une centaine de résidus, dont le repliement ne resemble à aucune autre. Cette approche marque une rupture avec les recherches développées jusque là, du côté de l’évolution dirigée ou de l’ingénierie protéique, où l’on partait d’une protéine existante dont on modifiait ou modulait l’activité avec des changements ciblés dans la séquence. Dans le CPD, on repart de zéro et l’on reconstruit une séquence complète. Comme le dit Baker lui même Si vous voulez bâtir un avion, vous n’allez pas pas modifier un oiseau. Et il lui a d’abord fallu comprendre les grands principes fondamentaux qui régissent le repliement des protéines avant de pouvoir générer de nouvelles séquence inédites.
Alors certes, TOP7 ne sait pas faire grand chose. Mais elle est la preuve que l’on peut concevoir des protéines qui n’existent pas dans la nature et remplissent un cahier des charges déterminé à l’avance (ici il s’agissait de sa structure en cinq feuillets et deux hélices). Les deux décennies qui ont suivi ont été incroyablement riches pour l’équipe de David Baker, qui a réussi à concevoir des protéines aux fonctions variées. Certaines sont capables de se lier à l’hémagglutinine de la grippe (afin de faciliter un diagnostique médical), de fixer des opioïdes (pour leur détection), ou encore d’empêcher une tempête de cytokines (et donc une réaction inflammatoire trop importante de notre organisme).
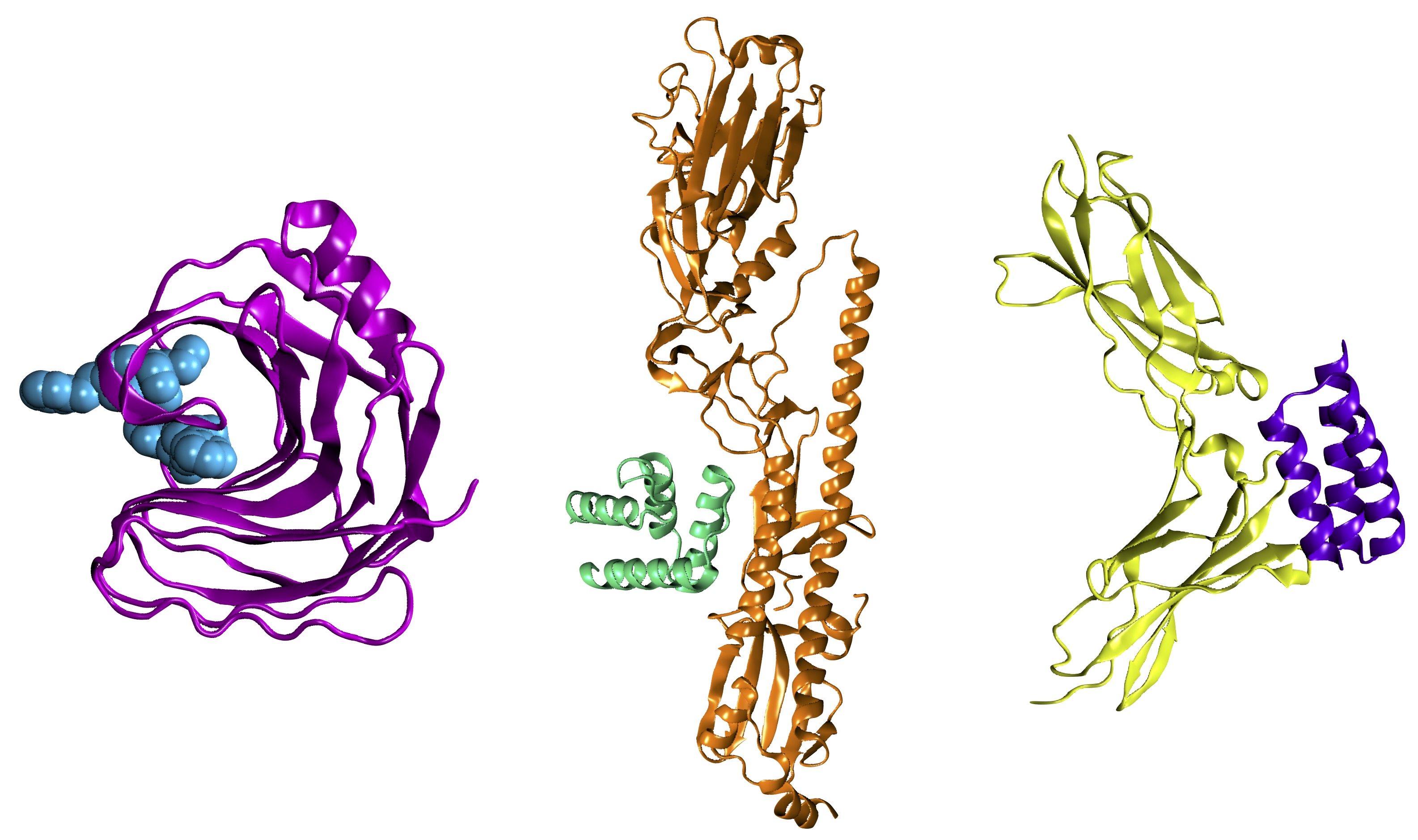
Avec la vingtaine de briques de base disponibles, pour des protéines qui comprennent souvent plusieurs centaines d’acides aminés, les possibilités en terme de conception de protéines sont infinies. L’ère du Computational Protein Design ne fait que commencer !
